Bioinformatik der Individualisierten Medizin

Forschungsfokus
Die Forschungsgruppe "Computational Biology for Individualized Infection Medicine" untersucht die Interaktion von genetischem Hintergrund und Umwelt und deren Beitrag zu Infektions- und immunbezogenen Krankheiten.
Wir konzentrieren uns auf die Anwendung und Entwicklung von computergestützten und statistischen Ansätzen zur Untersuchung der Auswirkungen genetischer Faktoren auf verschiedenen molekularen Ebenen (z.B. Genetik, Genomik, Metabolomik usw.), immunologischer Parameter und Funktionen, sowie auf komplexe Krankheiten.
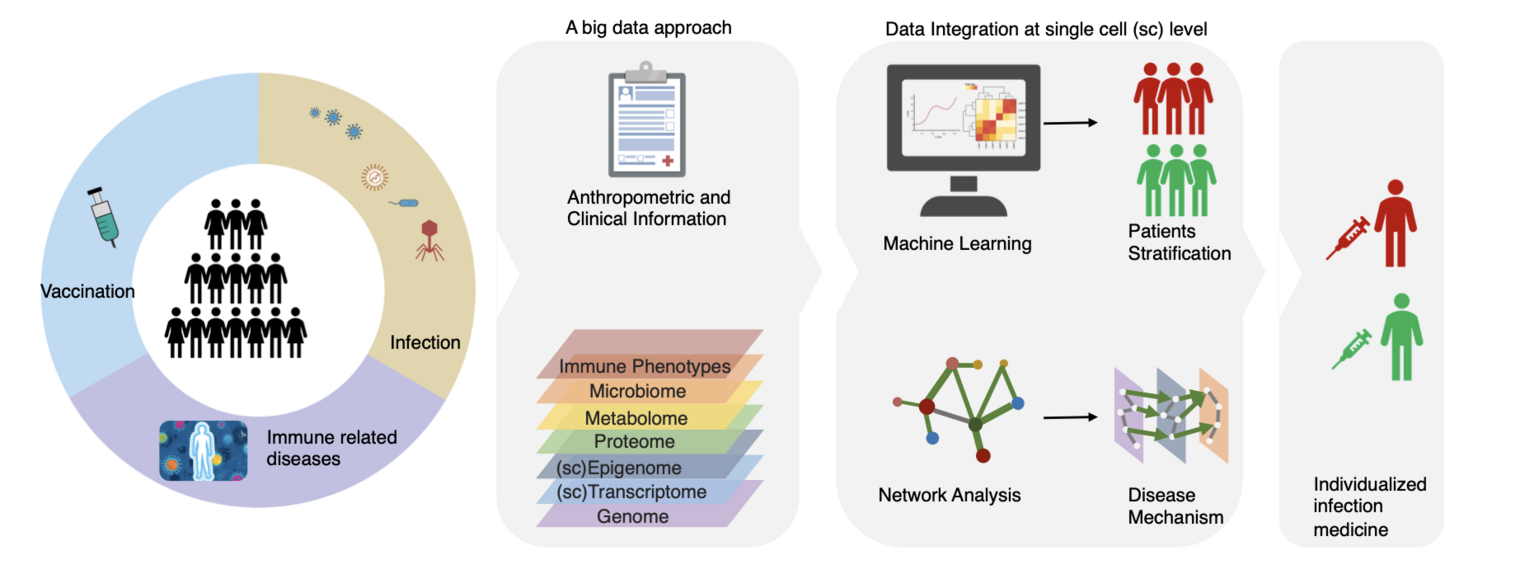
Unser Ziel ist es, die genetischen Risikofaktoren der Betroffenen und ihre zugehörigen molekularen Pfade aufzudecken und damit die Identifizierung von Risikopatienten zu verbessern. Diese Erkenntnisse sind für das Verständnis und die individualisierte Behandlung von Infektionskrankheiten entscheidend.

Forschungsfokus
Die Forschungsgruppe "Computational Biology for Individualized Infection Medicine" untersucht die Interaktion von genetischem Hintergrund und Umwelt und deren Beitrag zu Infektions- und immunbezogenen Krankheiten.
Wir konzentrieren uns auf die Anwendung und Entwicklung von computergestützten und statistischen Ansätzen zur Untersuchung der Auswirkungen genetischer Faktoren auf verschiedenen molekularen Ebenen (z.B. Genetik, Genomik, Metabolomik usw.), immunologischer Parameter und Funktionen, sowie auf komplexe Krankheiten.

Unser Ziel ist es, die genetischen Risikofaktoren der Betroffenen und ihre zugehörigen molekularen Pfade aufzudecken und damit die Identifizierung von Risikopatienten zu verbessern. Diese Erkenntnisse sind für das Verständnis und die individualisierte Behandlung von Infektionskrankheiten entscheidend.
Prof. Dr. Yang Li
Menschen unterscheiden sich in ihrem genetischen Hintergrund. Wir untersuchen diese interindividuelle Variation und wollen die Anfälligkeit für Infektionen verstehen und das Krankheitsrisiko vorhersagen

Yang Li ist eine Bioinformatikerin , die sich auf die Integration von Multi-Omics- und Einzelzell-Omics-Daten spezialisiert hat, um die Interaktionen zwischen dem genetischen Hintergrund und der Umwelt in Bezug auf Infektionen und immunbasierten Krankheiten zu untersuchen. Y. Li zeigte bereits, wie genetische Risikofaktoren der Betroffenen und ihre entsprechenden molekularen Eigenschaften die menschlichen Immunfunktionen bestimmen und vorhersagen, was für die individualisierte Behandlung von Infektionskrankheiten und Allergien entscheidend ist (Cell 2016, Nat Med 2016, Nat Immunol 2018). Kürzlich führte ihr Team die erste Multi-Omics-Studie zu dysfunktionalen Immunsystemen bei leichten und schweren COVID-19-Patienten durch (Cell 2020) und deckte dabei die epigenetischen und genetischen Einflüsse der angeborenen Immunität gegenüber COVID-19 auf (Cell Genomics 2023). Für ihre Forschung erhielt sie den renommierten ERC Starting Grant „ModVaccine“, zur Verbesserung von Impfstoffen (2020).
Prof. Dr. Li ist Mitglied im Redaktionsbeirat mehrerer angesehener wissenschaftlicher Zeitschriften, wie Frontiers in Immunology und Frontiers in Genetics. Weiterhin ist sie national und international als Gutachterin für die Deutsche Forschungsgemeinschaft (DFG), das Deutsche Zentrum für Infektionsforschung (DZIF), die Israel Science Foundation, die Swiss National Science Foundation, das Medical Research Council (UK) und die French Foundation for Medical Research tätig. Darüber hinaus ist sie Teil der Deutschen Gesellschaft für Immunologie, der Amerikanischen Gesellschaft für Humangenetik und der Europäischen Gesellschaft für Humangenetik. Außerdem ist sie Mitglied des Beirats und des Expertenpools des Konsortiums Milieu Intérieur des Institut Pasteur in Frankreich.
Forschungsprojekte
Wir nutzen einen Big-Data-Ansatz um Immunfunktionen bei Impfungen und Infektionen zu untersuchen. Hierbei konzentrieren wir uns insbesondere auf die folgenden Bereiche:
Integration von Multi-omics-Daten
Zur Entwicklung von Rechenmodellen, Erkennung von wichtigen biologischen Signalwegen und molekularen Wechselwirkungen, sowie Makern, welche mit Krankheiten und Eigenschaften assoziiert sind, nutzen wir einen Ansatz, der Multi-Omics-Datensätze und Immunprofile von Patienten-/Kontrollkohorten verwendet.
Single-cell genomics
Darüber hinaus entwickeln und verwenden wir modernste Technik zur Untersuchung der Heterogenität von Zellpopulationen, zur Identifizierung seltener Zelltypen und zum Verständnis der Zelldzynamik bei verschiedenen Krankheiten basierend auf der Untersuchung einzelner Zellen.
Genetische Regulierung des molekularen und immunologischen Phänotyps
Unsere Forschung konzentriert sich darauf zu verstehen, wie genetische Variationen molekulare und immunologische Phänotypen wie z.B. Genexpression, Metaboliten und Zytokinreaktionen auf Stimulationen beeinflussen. Hierfür entwickeln wir Berechnungsmethoden und Algorithmen, um High-throughput Datasets vollständig zu nutzen, welche die neuste Profilerstellungstechnologie wie z.B. kausaler Inferenz und Dekonvolution entstammen.
Insgesamt ermöglicht uns der Big-Data-Ansatz ein umfassendes Verständnis der Immunfunktionen bei Impfungen und Infektionen. Durch die Untersuchung der genetischen Grundlagen und molekularen Interaktionen, welche die Grundlage für komplexe Merkmale und Krankheiten bilden, hoffen wir, eine personalisierte Infektionsmedizin zu entwickeln, in der individuelle Unterschiede in der genetischen Variation und den Immunreaktionen berücksichtigt werden können.
In den letzten Jahren hat unsere Forschung einen Beitrag zu den folgenden vier Hauptbereichen geleistet:
Infektionskrankheiten
Anhand von Transkriptom-, Genom- und Epigenomdaten von hospitalisierten und rekonvaleszenten COVID-19-Patienten konnten wir zeigen, dass eine veränderte Chromatin-Zugänglichkeit und allelspezifisches, offenes Chromatin mit einer gestörten epigenetischen Regulierung einhergeht, die zur Pathogenese von COVID-19 beiträgt (Cell Genomics 2023). Mit Hilfe von Multi-Omics-Ansätzen für einzelne Zellen konnten wir geringfügige Unterschiede in den epigenetischen und transkriptionellen Programmen des Immunsystems von COVID-19-Patienten nachweisen, welche sich jedoch größtenteils auf ein normales Niveau zurück entwickeln. Allerdings ist das zirkulierende Proteom, bei Post-COVID-19 Patienten, selbst mehrere Wochen nach der Infektion noch beeinträchtigt (Frontiers in Immunol. 2022).
Immunkrankheiten
Durch die Untersuchung dynamischer Transkriptionsveränderungen in einzelnen Zellen pädiatrischer Personen, konnten wir mit Zöliakie assoziierte, unterschiedlich ausgedrückte Gene und genetische Risikoloci aufdecken (Frontiers in Immunol. 2022). Darüber hinaus haben wir in einer Studie zur funktionellen Genomik bei T1D neue Erkenntnisse über die genetischen Faktoren gewonnen, die immunologischer Reaktionen bei T1D beeinflussen (eLife 2022).
Zusammen mit den Klinikern der MHH haben wir eine Einzelzell-Multiomics-Studie zu atopischer Dermatitis (AD) und Psoriasis (PS) durchgeführt und festgestellt, dass die krankheitsspezifischen T-Zell-Cluster bei AD hauptsächlich zu einer TH2/Th22-Subpopulation und bei PS zu einer TH17/tc17-Subpopulation gehörten und ihre Anzahl in beiden Krankheiten mit dem Schweregrad der Krankheit zusammenhing.
Impfreaktion
Mithilfe eines integrativen genomischen Ansatzes in zwei unabhängigen bevölkerungsbasierten BCG-Impfkohorten gesunder Personen konnten wir genetische Loci nachweisen, die die Zytokinantworten der trainierten Immunität, sowie die H3K9-Histodemethylasen der KDM4-Familie als ihre wichtigen Regulatoren beeinflussen (Eur. J. Immunol. 2022). Weiterhin präsentierten wir einen umfassenden überblick über die zellulären Transkriptionsprogramme der trainierten Immunität bei einer Einzelzellauflösung und zeigten somit die Heterogenität der Genexpression in trainierten Zellen auf (J. Clint. Invest 2022). Zusätzlich haben wir festgestellt, dass zirkulierende Metaboliten wichtige Faktoren sind, die die BCG-induzierte trainierte Immunität beim Menschen beeinflussen und dass die Modulation von Stoffwechselwegen eine neuartige Strategie zur Verbesserung der Impfstoff- und trainierten Immunitätsreaktionen sein könnte (PLOS Biol 2022).
Entwicklung neuer Berechnungsmethoden
Wir haben gezeigt, dass eQTL-Dekonvolutionsanalysen ein leistungsfähiges Instrument zum Verständnis zelltypspezifischer molekularer Mechanismen der Asthmaentwicklung sein können (Allergy 2022).












Ausgewählte Publikationen
1. Chu, X., Janssen, A.W.M., Koenen, H., Chang, L., He, X., Joosten, I., Stienstra, R., Kuijpers, Y., Wijmenga, C., Xu, C.- J., Netea, M.G, Tack, C.J., Li, Y.# A genome-wide functional genomics approach uncovers genetic determinants of immune phenotypes in type 1 diabetes. eLife, 11, e73709 (2022). https://doi.org/10.7554/eLife.73709
2. Koeken, V.A.C.M., Qi, C., Mourits, V.P., de Bree, L.C.J., Moorlag, S.J.C.F.M., Sonawane, V., Heidi Lemmers, Dijkstra, H., Joosten, L.A.B., van Laarhoven, A., Xu, C.-J., van Crevel, R., Netea, M.G., Li, Y.# Plasma metabolome predicts trained immunity responses after antituberculosis BCG vaccination. PLoS Biol20(9): e3001765, (2022). https://doi.org/10.1371/journal.pbio.3001765
3. Qi, C., Berg, M., Chu, X., Van Den Berge, M., Xu, C., Koppelman, G., Nawijn, M., Li, Y. Cell type eQTL deconvolution of bronchial epithelium through integration of single cell and bulk RNA-seq. Allergy 77, 3663-3666 (2022).https://doi.org/10.1111/all.15410
4. Zhang, B., Moorlag, S., Domínguez-Andrés, J., Bulut, Ö., Kilic, G., Liu, Z., van Crevel, R., Xu,C.-J., Joosten, L.A.B., Netea, M.G.#, Li, Y.# Single-cell RNA sequencing reveals induction of distinct trained immunity programs in human monocytes. J Clin Invest, 132(7), e147719 (2022) https://doi:10.1172/JCI147719
5. Zhang, B, Roesner, L.M., Traidl, S., Koeken, V.A.C.M., Xu, C.-J., Werfel, T., Li, Y. Single-cell profiles reveal distinctive immune response in atopic dermatitis in contrast to psoriasis. Allergy, 00: 1- 15 (2022). https://doi: 10.1111/all.15486
Eine vollständige Liste der Publikationen von Yang Li finden Sie bei GoogleScholar.
Jobs
Die Gruppe "Bioinformatik der Individualisierten Medizin" freut sich jederzeit über Bewerbungen von qualifizierten Kandidaten für eine Promotion oder eine Postdoc-Stelle.
Alle offenen Stellenausschreibungen finden Sie auf der Jobportal-Seite des HZI.
Initiativbewerbungen sind zudem jederzeit willkommen. Wir sind auf der Suche nach motivierten Bewerbern mit einem starken Hintergrund in Bioinformatik / Computerbiologie, Informatik, Statistik, Biologie oder Physik, guten Programmierkenntnissen und Interesse an interdisziplinärer Forschung in der Biologie und Infektionsforschung.
Weiterhin bieten wir kontinuierlich Master-/Bachelor-Forschungsprojekte an. Bitte kontaktieren Sie uns für weitere Informationen per E-Mail.
Unsere Forschung konzentriert sich auf das Verständnis interindividueller Variationen in der Anfälligkeit für immungeschwächte Erkrankungen und die Vorhersage von Immunfunktionen durch die Integration großer Multiomics und detaillierter Datensätze von Immunphänotypen aus einer Vielzahl von bestehenden und geplanten Patientenkohorten. Methoden, die wir in unserer Forschung anwenden oder entwickeln, beziehen sich auf die Bereiche Bioinformatik, Systemgenetik, Machine Learning & Deep Learning und Systemimmunologie.
Bitte senden Sie - soweit in einer konkreten Stellenauschreibung nicht anders angegeben - Ihre Bewerbungen an Yang Li. Bitte beachten Sie hierfür unsere Datenschutzerklärung.
Weitere Informationen
Entwicklung neuer Berechnungsmethoden
Da Studien auf der Grundlage von Massen-RNA-seq-Daten durch die fehlende Zelltyp-Auflösung eingeschränkt sind, haben wir einen eQTL-Dekonvolution-Ansatz entwickelt, um zelltypspezifische genetische Effekte der Genexpression krankheitsassoziierter Varianten zu identifizieren (BMC Bioinfo. 2020). Unsere Ergebnisse konnten mit der Einzelzell-eQTL-Methode repliziert werden, was darauf hindeutet, dass eQTL-Dekonvolution-Analysen ein leistungsfähiges Instrument zum Verständnis zelltypspezifischer molekularer Mechanismen der Asthmaentwicklung sein können (Allergy 2022). In meinem Labor wurde ein Demultiplexing-Analyserahmen entwickelt, der die künftige Analyse von Einzelzell-omics-Datensätzen verbessern wird (Zoodsma et al., in Vorbereitung).
Details als auch Modelle finden Sie hier.